En la madrugada del día 03 de Octubre de este año, George Pearson Smith recibió la noticia de que había sido uno de los ganadores del premio Nobel de Química. Su primera reacción fue pensar que era una de las tantas bromas que recibió por esos días, sin embargo esa llamada en particular era real, lo supo por la interferencia (estática) de fondo. Pues bien, George Smith es un científico estadounidense nacido en Connecticut en el año 1941, formado en Biología y doctor en Bacteriología e Inmunología por la Universidad de Harvard. Después de algunos años de posdoctorado trabajando bajo la supervisión de Oliver Smithies, también ganador de un premio Nobel en Fisiología / Medicina, Smith se incorporó como profesor de la Universidad de Missouri, donde permanece hasta la fecha en calidad de profesor emérito de Ciencias Biológicas. La Real Academia Sueca de Ciencias decidió otorgarle el Nobel de Química, que compartió con los científicos Frances Arnold y Gregory Winter, por su trabajo en el desarrollo de la técnica de Phage Display (“presentación en fagos”).

En 1985 George Smith publicó un artículo científico en la prestigiosa revista Science titulado “Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface”, (http://science.sciencemag.org/content/228/4705/1315/tab-pdf), el cual representó el nacimiento de la técnica de Phage Display, que revolucionaría diversos campos de la biología. En este artículo Smith presentaba un método elegantemente diseñado y de extrema versatilidad, en el cual se utilizan virus que infectan bacterias (bacteriófagos) para expresar proteínas de diversas conformaciones en su superficie. Más o menos sería como comenzar a “programar” virus para que éstos produzcan moléculas de interés científico, convirtiéndolos en nuestros aliados en la ciencia.
Inicialmente, Smith trabajó diseñando y expresando proteínas de fusión a través de la inserción de fragmentos de DNA unidos al gen pIII de bacteriófagos M13, de esa forma los fragmentos insertados podían ser expresados (presentados) en la superficie de los virus modificados genéticamente, unidos a la proteína pIII, sin afectar su capacidad de infección ni su viabilidad. A partir de su publicación esta técnica revolucionó el proceso de descubrimiento de moléculas con características biológicas altamente específicas (deseables), favoreciendo la identificación de péptidos o fragmentos de anticuerpos que pudieran ser utilizados en áreas como diagnóstico de enfermedades, rastrear marcadores biológicos de cáncer, detectar toxinas y un sinnúmero de aplicaciones de interés biotecnológico y de salud pública. Desde su aparición en 1985, la aplicación de esta técnica ha permitido la publicación de millares de artículos científicos y un gran número de patentes en las más diversas áreas de las ciencias. En la figura 1 se puede observar de forma resumida el proceso de Phage Display descrito inicialmente por Smith (1985).
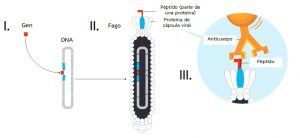
La elegancia experimental de la técnica de Phage Display reside en que en cada fago utilizado existe una robusta relación genotipo-fenotipo (ADN-proteína), la cual posibilita que a través de una técnica relativamente sencilla como la secuenciación de ADN del fago sea posible definir la secuencia del péptido que está expreso en la superficie de cada virus, permitiendo su posterior producción y optimización a través de diversas técnicas de ingeniería genética.
La construcción de bibliotecas de Phage Display (phage display libraries) ha permitido la selección de una enorme cantidad de moléculas con características biológicas y estructurales altamente específicas, a partir de grandes colecciones de virus expresando péptidos, proteínas o fragmentos de anticuerpos (bibliotecas). Uno de los primeros grandes avances en esta área fue la aleatorización de las secuencias de los péptidos presentados en la superficie de los fagos, permitiendo de esa forma la construcción de bibliotecas de fagos que pueden llegar a contener varios billones de péptidos diferentes. Por otro lado, esta técnica también ha sido adaptada para permitir la expresión de grandes repertorios de anticuerpos diferentes dirigidos contra la misma molécula, haciendo posible la posterior selección, a través de rounds de biopanning (ver figura 2), de anticuerpos con características altamente específicas para los investigadores. Además, es necesario señalar que la construcción de bibliotecas de Phage Display ha hecho posible la experimentación con billones de péptidos o fragmentos de anticuerpos en experimentos únicos, bajo condiciones experimentales relativamente fáciles de controlar en el laboratorio.
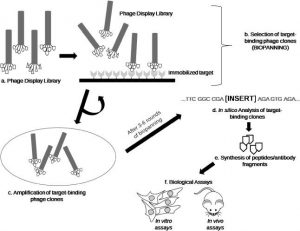
De acuerdo con diversos científicos, la aplicabilidad del método de Phage Display podría estar limitada apenas por la imaginación de los investigadores, debido a que su constante uso por la comunidad científica la ha convertido en un método robusto, altamente versátil, potente y adaptable a numerosas condiciones experimentales. Éstas características la convierten en una herramienta poderosa para la generación de conocimiento que pudiera conducir a encontrar soluciones de interés científico, las cuáles pudieran ser revertidos en beneficio de la humanidad.
Información adicional:
Reviews Phage Display:
- https://www.tandfonline.com/doi/full/10.1080/1061186X.2016.1258570 (Rahbarnia et al., 2017)
- https://www.sciencedirect.com/science/article/pii/S0041010116300277?via%3Dihub#fig2 (Bernedo-Navarro & Yano, 2016)
Video: https://www.youtube.com/watch?v=VgDF-N-73U0

Este Articulo fue escrito por Robert Alvin Bernedo Navarro, microbiólogo peruano, doctor en Biología Funcional y Molecular por la Universidade Estadual de Campinas (UNICAMP). El autor ha publicado artículos científicos en el área de Phage Display tanto de péptidos como de anticuerpos durante su doctorado y sus estancias post doctorales en la UNICAMP (Brasil) y la Vrije Universiteit Brussel (VUB – Bélgica).


![[Artículo de opinión] Por una sociedad peruana que mire a la ciencia con confianza y respeto](https://www.cientificos.pe/wp-content/uploads/2020/04/170422114451-13-march-for-science-0422-nyc-exlarge-169-218x150.jpg)